CRISPR-Cas9基因编辑工具在人类细胞基因编辑研究和基因治疗领域有广泛的应用前景。随着对这项技术研究的不断深入,科学家发现CRISPR-Cas9介导双链DNA断裂,细胞进行DNA修复后,除了可能在双链DNA断裂位置产生短的插入缺失突变,还有可能产生大片段的DNA缺失突变和杂合子丢失[1-4]。这些大片段缺失突变的长度能够达到几百bp至数千bp以上,在临床基因治疗中可能存在安全风险,因此需要对基因编辑细胞产品的大片段缺失突变进行检测,并开发相关技术进一步规避或降低大片段缺失突变的产生。
2021年8月20日,中国医学科学院血液病医院(中国医学科学院血液学研究所)张孝兵/程涛团队在《Genome Biology》上在线发表题为“Effective control of large deletions after double-strand breaks by homology-directed repair and dsODN insertion”的论文。

该论文系统研究了人原代T细胞、造血干祖细胞和诱导多能性干细胞在CRISPR-Cas9基因编辑后产生的大片段缺失突变(图1),比较并提出多种包括HDR或者NHEJ介导的基因编辑方案来降低基因编辑后大片段缺失突变的产生。
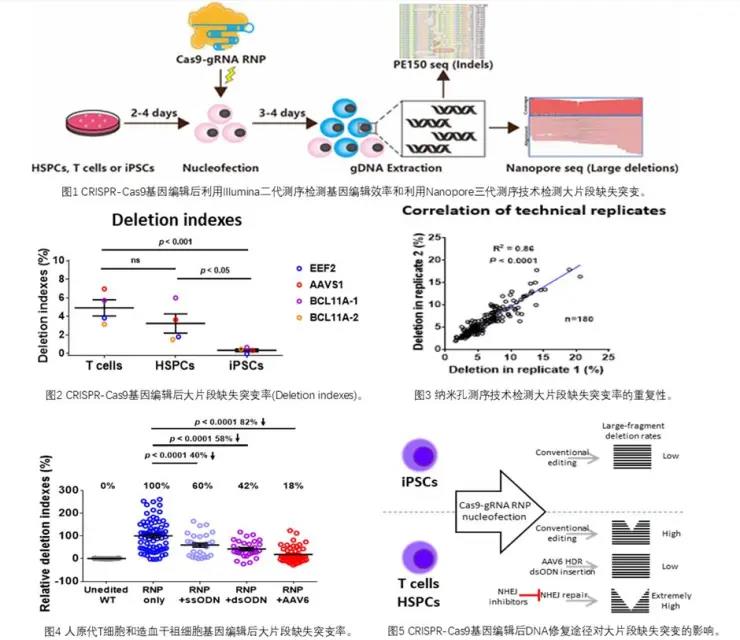
作者对临床相关的人类细胞进行基因编辑,利用长片段PCR和纳米孔测序技术,检测了多个基因编辑位点的大片段缺失突变(图2)。结果发现,人原代T细胞基因编辑后产生较高(3-7%)的大片段缺失突变率,造血干祖细胞居中(2-6%),而人诱导多能性干细胞的大片段缺失突变几率很低(<1%)。为了佐证结果的可靠性,作者将不同barcode标记相同靶向引物的180个技术重复组进行关联分析,发现大片段缺失突变具有很好的相关性(R2=0.86,P<0.0001)(图3)。此外,作者进一步分析了52个基因编辑的人诱导多能性干细胞单细胞克隆,发现只有一个克隆在一条染色体上存在482-bp的大片段缺失突变,所有克隆都没有出现杂合子丢失。
CRISPR-Cas9基因编辑时提供同源重组模板(如ssODN或AAV)能实现定点基因敲入。作者发现,使用ssODN实现基因敲入,使大片段缺失突变率降低50%,使用AAV载体作为同源重组模板时,大片段缺失率降低80%。还发现,同源重组模板不会影响T细胞或者造血干祖细胞的基因编辑效率。因此,HDR介导的基因修复能够有效降低T细胞和造血干祖细胞基因编辑后,大片段缺失突变的产生(图4)。
作者还探索了在不存在HDR模板的情况下,怎样降低突变率。由于Cas9-sgRNA产生双链DNA断裂后,NHEJ修复途径可以介导短的双链DNA (dsODN)的插入,这一方法已经用来检测基因编辑后的脱靶效应[5]。作者发现,在T细胞和造血干祖细胞中,CRISPR-Cas9基因编辑技术能实现dsODN(28-34bp)的插入且不会影响基因编辑效率,但能够有效降低基因编辑后大片段缺失突变率约60%(图4)。当添加NHEJ抑制剂M3814时,dsODN插入效率降低90%,提示dsODN插入依赖于NHEJ修复途径。当添加NHEJ抑制剂时,基因编辑后大片段缺失突变率显著提高,说明抑制NHEJ修复途径能增加大片段缺失突变。因此,结合HDR介导的基因编辑的结果,作者认为,HDR和NHEJ修复途径在减少基因编辑后大片段缺失突变起到了至关重要的作用(图5)。
这项研究综合阐述了CRISPR-Cas9基因编辑后大片段缺失突变的检测方法,提出HDR和NHEJ介导的多种基因编辑方案,能够有效降低基因编辑后大片段缺失突变概率,填补了目前尚无有效方案解决大片段缺失突变的技术空白。目前,人原代T细胞和造血干祖细胞已经广泛应用于人类疾病的临床研究和基因治疗,其基因编辑后产生的较高的大片段缺失突变率应当引起科学家和临床医生的关注,该项研究成果可以为今后基因治疗方案的设计提供参考和技术支持。
中国医学科学院血液病医院(中国医学科学院血液学研究所)、实验血液学国家重点实验室张孝兵研究员和程涛教授为本文的共同通讯作者,人诱导多能干细胞技术平台负责人温伟、硕士研究生权子莙和硕士研究生李斯昂为本文的共同第一作者。该研究在国家自然科学基金委、科技部国家重点研发计划和中国医学科学院医学与健康科技创新工程等基金支持下完成。
参考文献:
1. Adikusuma, F., et al., Large deletions induced by Cas9 cleavage. Nature, 2018. 560(7717): p. E8-E9.
2. Kosicki, M., K. Tomberg, and A. Bradley, Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nat Biotechnol, 2018. 36(8): p. 765-771.
3. Weisheit, I., et al., Detection of Deleterious On-Target Effects after HDR-Mediated CRISPR Editing. Cell Rep, 2020. 31(8): p. 107689.
4. Zuccaro, M.V., et al., Allele-Specific Chromosome Removal after Cas9 Cleavage in Human Embryos. Cell, 2020.
5. Tsai, S.Q., et al., GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol, 2015. 33(2): p. 187-197.
撰稿:权子莙 李斯昂
编辑:董源
校对:徐龙彪
审核:祁健伟 周家喜 程 涛