细胞内的动态过程主要受基因之间相互作用的调控,而基因互作网络的紊乱可能会改变细胞的状态或功能,进而引发肿瘤等复杂疾病的发生[1,2]。构建基因互作网络有助于我们更好地理解成千上万的基因是如何相互作用的,对于深入解析疾病发生发展机制至关重要[3]。然而,基因互作网络可能会在不同的时间和空间背景下发生重塑[4]。随着单细胞RNA测序技术的不断进步,现在已能够在单细胞水平上解析异质性组织。因此,利用单细胞转录组数据来构建细胞类型特异性的基因互作网络,有助于研究疾病基因的细胞类型特异性效应。
近日,中国医学科学院血液病医院(中国医学科学院血液学研究所)原佳沛研究员与天津医科大学杨扬教授合作在《Genome Medicine》(IF=12.3)发表了题为“An atlas of cell-type-specific interactome networks across 44 human tumor types”的研究论文。该研究汇总了44种不同肿瘤类型的单细胞转录组数据,构建了肿瘤中不同细胞类型特异性的基因互作网络图谱和泛癌种免疫微环境中不同免疫细胞亚型特异性的基因互作网络图谱,并建立了可供用户在线检索、下载及分析的平台CellNetdb,为研究细胞类型特异性相互作用提供多种方法。

首先,研究发现细胞类型特异性基因互作网络在识别肿瘤中细胞类型特异性基因方面发挥着重要作用。以泛癌免疫微环境中的髓系细胞为例,研究表明已知的特征基因在相应的细胞类型中展现出明显更高的拓扑结构特异性。通过对基因的拓扑结构特异性和表达水平特异性评分进行比较,该研究成功鉴定出一些基因,它们在多个细胞亚群中均有表达,但在细胞类型特异性互作网络中呈现出不同的拓扑结构特征,如调节巨噬细胞炎症的CD74和参与肌动蛋白结合的CAP1。结果表明,细胞类型特异性网络的建立有助于补充传统基于基因表达模式的分析方法,从基因互作网络层面解释细胞类型特异性。
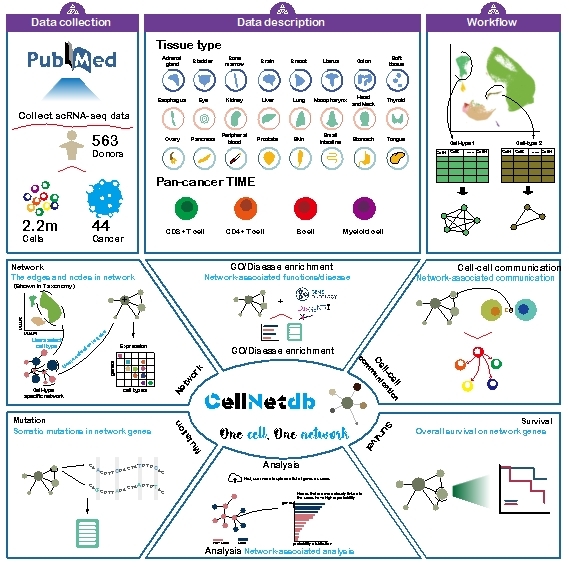
然后,该研究通过分析预后基因集合在不同细胞类型特异性基因互作网络中的连接度,将预后基因集合与不同细胞类型联系起来,揭示了与肿瘤预后相关的特定细胞类型。分析结果显示,在许多非恶性细胞的特异性基因互作网络中,预后基因集合呈现出显著的网络紧密性,表明一些预后基因可能通过肿瘤微环境中的非恶性细胞发挥作用,如胃癌中的成纤维细胞。进一步研究发现,在胃癌的成纤维细胞特异性互作网络中,预后基因ITGB1具有最高的网络中心度,与ITGB1直接相连的基因包括多个预后基因,如与肿瘤成纤维细胞相关的EGFR、FN1及COL1A1,表明ITGB1的预后效应与肿瘤相关成纤维细胞介导的肿瘤进展密切相关。研究结果显示,肿瘤中细胞类型特异性互作网络能够将预后效应关联到特定的细胞类型,并有助于识别特定细胞类型中潜在的关键靶点。
此外,该研究利用重启随机游走算法,建立了基于细胞类型特异性基因互作网络的风险基因排优化方法,可以在特定细胞类型背景下识别肿瘤相关风险基因。该方法在葡萄膜黑色素瘤和急性髓细胞白血病中的成功应用证明了其在肿瘤驱动基因排优化方面的有效性。该研究还将该方法应用于耗竭性T细胞特异性互作网络中,鉴定出了一些以前未报道的T细胞耗竭相关基因,并结合肿瘤免疫检查点阻断治疗队列数据对其进行了阐明,有助于为恶性肿瘤的诊疗提供新的潜在靶点。
天津医科大学基础医学院杨扬教授和中国医学科学院血液病医院(中国医学科学院血液学研究所)原佳沛研究员为本文的通讯作者,李泽昆、刘格睿和杨晓晓为本文的共同第一作者。该项目获得国家自然科学基金、中国医学科学院医学与健康科技创新工程等基金的支持。
参考文献
1. Sahni N, Yi S, Taipale M, Fuxman Bass JI, Coulombe-Huntington J, Yang F, et al. Widespread macromolecular interaction perturbations in human genetic disorders. Cell. 2015;161(3):647-60.
2. Vidal M, Cusick ME, Barabasi AL. Interactome networks and human disease. Cell. 2011;144(6):986-98.
3. Szklarczyk D, Gable AL, Lyon D, Junge A, Wyder S, Huerta-Cepas J, et al. STRING v11: protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets. Nucleic Acids Res. 2019;47(D1):D607-D13.
4. Scott JD, Pawson T. Cell signaling in space and time: where proteins come together and when they're apart. Science. 2009;326(5957):1220-4.